Detection of enhancer activity at the single-cell level by deep learning method
Authors:Murakami Ken
Affiliation:The University of Osaka Graduate School of Medicine
Abstract:
目的:組織は多種多様な細胞が環境に応答し、それぞれ異なる遺伝子発現を示すことによって機能している。そのためにはエンハンサーとよばれる遺伝子発現の制御領域が細胞ごとに異なる活動を示す必要がある。しかし現在の手法ではエンハンサー領域を一細胞レベルで同定することは不可能である。そこで深層学習手法とシングルセルデータから一細胞レベルでエンハンサー領域を決定する手法を開発する。
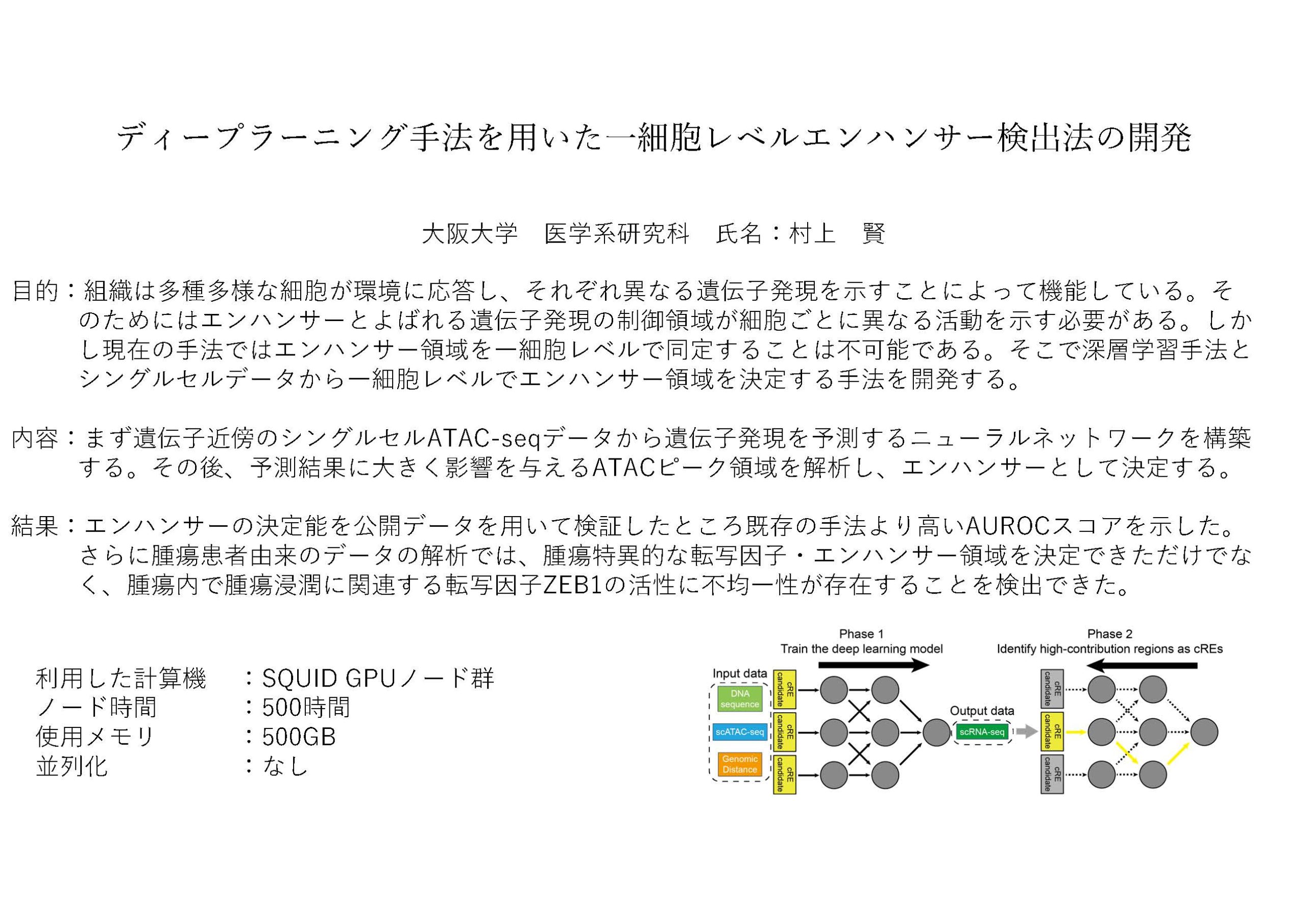
内容:まず遺伝子近傍のシングルセルATAC-seqデータから遺伝子発現を予測するニューラルネットワークを構築する。その後、予測結果に大きく影響を与えるATACピーク領域を解析し、エンハンサーとして決定する。
結果:エンハンサーの決定能を公開データを用いて検証したところ既存の手法より高いAUROCスコアを示した。さらに腫瘍患者由来のデータの解析では、腫瘍特異的な転写因子・エンハンサー領域を決定できただけでなく、腫瘍内で腫瘍浸潤に関連する転写因子ZEB1の活性に不均一性が存在することを検出できた。
Publication related to your research:
(Journal paper)
- Ken Murakami, Keita Iida, Mariko Okada, "An Attention-Based Deep Neural Network Model to Detect Cis-Regulatory Elements at the Single-Cell Level From Multi-Omics Data.", Genes to cells, Vol. 30, No. 2, e70000, Mar. 2025
Posted : March 31,2025